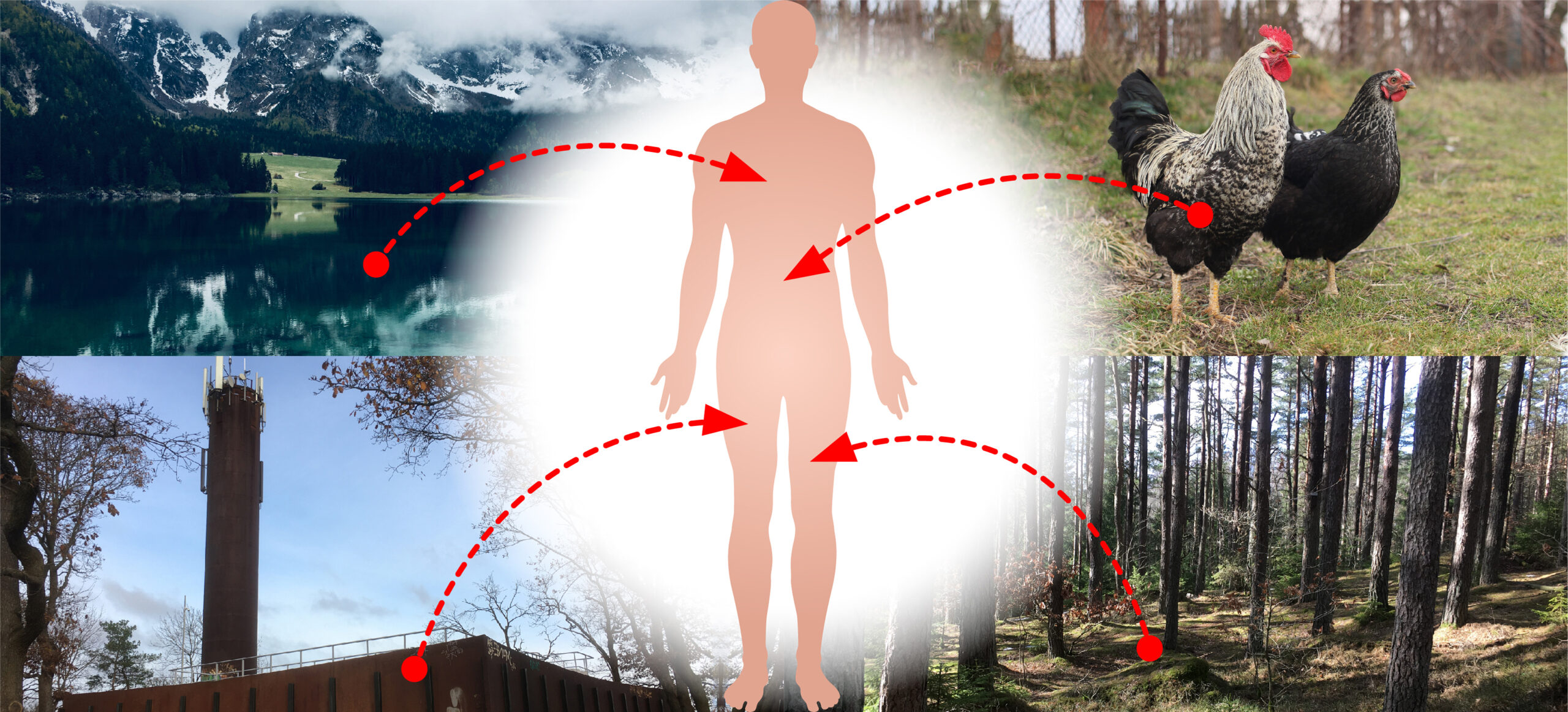
Bakterier hos människan utvecklar idag antibiotikaresistens betydligt snabbare än vad man kunnat räkna med.
 Nu visar teoretisk forskning på Chalmers att en orsak skulle kunna vara en omfattande överföring av gener mellan bakterier i våra ekosystem och till människan. Arbetet har också lett till nya verktyg för resistensforskare.
Nu visar teoretisk forskning på Chalmers att en orsak skulle kunna vara en omfattande överföring av gener mellan bakterier i våra ekosystem och till människan. Arbetet har också lett till nya verktyg för resistensforskare.
Antibiotikaresistens är enligt WHO ett av de största hoten mot global hälsa, livsmedelssäkerhet och utveckling. Redan idag orsakar det över 33 000 dödsfall varje år enbart i Europa.
Helt olika bakteriearter kan sprida resistensgener till varandra med hjälp av så kallade plasmider. Det är friliggande små DNA-ringar där bakterier förvarar en del av sina gener utanför kromosomen. När två bakterieceller kommer i kontakt kan de kopiera över plasmider till varandra. Detta kallas för konjugation, och är den viktigaste mekanismen för spridning av antibiotikaresistens.
– Under senare år har det visat sig att resistensgener sprider sig till människans bakterier i mycket större omfattning än vad man hade kunnat vänta sig, säger Jan Zrimec, forskare i systembiologi på Chalmers. Många av generna verkar ha sitt ursprung i vitt skilda bakteriearter och miljöer, så som bakterier i jord, vattenmiljöer och växter.
– Detta har varit svårt att förklara, för även om konjugation är väldigt vanligt så har man trott att det ändå fanns en kraftfull begränsning för vilka bakteriearter som kan skicka plasmider till varandra. Plasmider tillhör olika så kallade mobilitetsgrupper, och kan därför inte överföras mellan vilka bakteriearter som helst.
Specifika DNA-regioner avslöjar spridningsmöjligheter
Jan Zrimec har utvecklat nya dataanalytiska metoder som visar att genöverföringen kan vara mycket mer gränslös och utbredd än vad man hittills har räknat med.
Bland annat har han tagit fram en algoritm som kan identifiera specifika DNA-regioner som behövs för konjugation – så kallade oriT – i stora datamängder som består av genuppsättningar från tusentals plasmider. Algoritmen kan dessutom sortera in plasmiderna i mobilitetsgrupper utifrån de oriT som identifieras.
Han har använt algoritmen för att utforska kända genuppsättningar hos drygt 4600 naturliga plasmider från olika typer av bakterier, något som tidigare inte har varit möjligt att göra på ett systematiskt sätt. Resultaten visar att:
- Antalet oriT kan vara nästan åtta gånger fler än de som hittas med den standardmetod som används idag.
- Antalet mobila plasmider kan vara dubbelt så många som man hittills har känt till.
- Antalet bakteriearter som har mobila plasmider kan vara nästan dubbelt så många som man hittills har känt till.
- Över hälften av dessa plasmider har oriT som passar ihop med ett konjugationsenzym från en annan plasmid som hittills har klassats till en annan mobilitetsgrupp. De skulle därför kunna överföras med hjälp av en sådan plasmid som råkar befinna sig i samma bakteriecell.
Det sista betyder att det kan finnas överföringsmekanismer mellan mängder av bakteriearter och miljöer där man hittills har trott att det fanns barriärer.
– Resultaten kan tolkas som att det finns ett robust nätverk för överföring av plasmider mellan bakterier i bland annat människor, djur, växter, jord, vattenmiljöer och industrier, säger Jan Zrimec. Resistensgener förekommer naturligt hos många olika bakterier i ekosystemen, och det hypotetiska nätverket skulle kunna innebära att gener från alla dessa miljöer kan överföras till bakterier som orsakar sjukdomar hos människan.
– Detta kan vara en möjlig orsak till den snabba resistensutvecklingen hos människans bakterier som har observerats under senare år. Vår stora antibiotikaanvändning selekterar fram resistensgenerna, som alltså skulle kunna flöda in från en mycket större naturlig genreservoar än vad man hittills har räknat med.
Kan göra bred nytta för bekämpning av antibiotikaresistens
Resultaten behöver verifieras av experimentella forskare framöver. Men de dataanalytiska metoder som Jan Zrimec har utvecklat är redan nu användbara för många av de forskare som arbetar med antibiotikaresistens inom olika medicinska och biologiska fält. De ger ett kraftfullt nytt verktyg för att systematiskt kunna kartlägga den möjliga spridningsförmågan hos olika plasmider.
– Detta har utgjort en stor begränsning för forskningsfältet hittills, säger Jan Zrimec. Jag hoppas att metoderna ska komma till nytta inom stora delar av forskningen om antibiotikaresistens, som är ett väldigt tvärvetenskapligt och fragmenterat område. Metoderna kan användas till studier för att ta fram både effektivare begränsningar för antibiotikaanvändning, instruktioner för hur antibiotika ska användas, och nya typer av substanser som kan hindra spridning av resistensgener på molekylär nivå.